(Situation au 8 avril au soir)
(mise-à-jour le 14 mars à midi) On peut trouver la suite de cet article ici, où une analyse de la robustesse du dernier algorithme est présentée.
Modélisations de l’évolution du nombre de cas de CoViD-19 en France
On présente ici deux modélisations possibles de l’évolution du nombre de cas de CoViD-19 en France.
La première consiste à modéliser l’évolution du nombre de cas par une exponentielle. Cette modélisation est une approximation assez efficace en début d’épidémie, mais ne peut pas être valable sur le long terme : en effet, une telle modélisation donne un nombre de cas qui tend vers $+\infty$ lorsque le temps passe, ce qui ne peut évidemment pas être valide dans un pays à population finie.
La seconde consiste à modéliser l’évolution du nombre de cas par une sigmoïde : $N_T=\dfrac{c}{1+e^{-(aT+b)}}$. Cette modélisation est plus délicate à obtenir mais présente l’avantage qu’elle pourrait se montrer valable sur le long terme.
On fournit ici et là les deux codes Python permettant d’obtenir les graphiques et les résultats envisagés pour ces deux simulations.
Ces simulations sont évidemment à prendre avec beaucoup de prudence et de circonspection !
Un graphique mis à jour se situe en fin d’article.
Enfin - ce sera ma dernière mise-à-jour de ce post - les évolutions des derniers jours (du 11 au 14 mars) semblent montrer que l’algorithme que j’utilisais minimisait largement l’ampleur de l’épidémie. Je publie en toute fin de post un nouvel algorithme où les paramètres de la régression linéaire sont pondérés par le nombre de cas détectés. Testé sur les données des jours précédents, cet algorithme se montre beaucoup plus robuste.
Régression exponentielle
On trouvera ci-dessous un code Python permettant de faire une régression linéaire du logarithme du nombre de cas de CoViD-19 $\ln(N_T)$ en fonction de la date $T$ : autrement dit, on cherche deux constantes réelles $a,b$ telles que la courbe d’équation $\ln(N_T)=aT+b$ minimise la somme des carrés des résidus entre le modèle choisi et les données publiées.
Cette régression repose sur un modèle - valable au début de l’épidémie seulement - dans lequel on assimile le nombre $N_T$ de cas à une fonction exponentielle du temps $T$ :
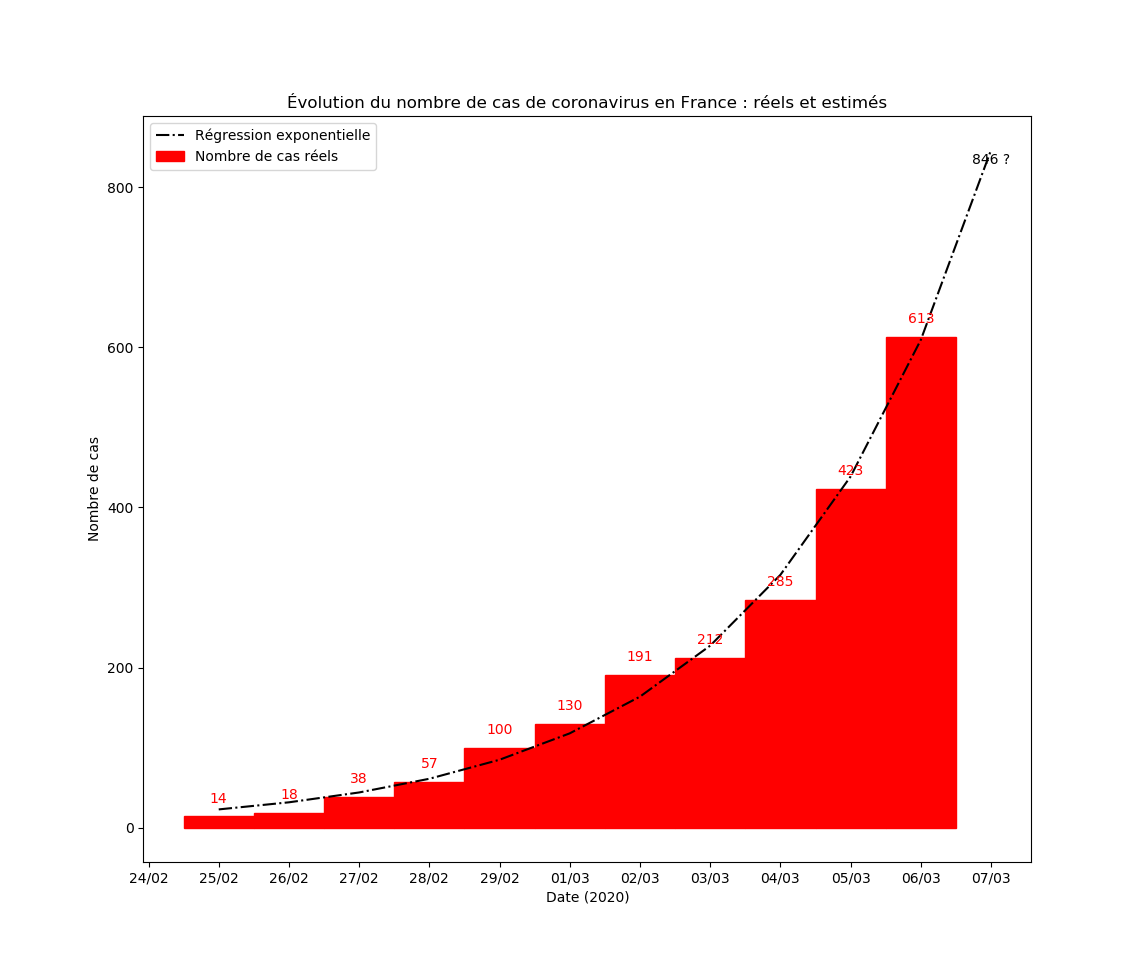
\[N_T=e^be^{aT}=\alpha C^T\]Ici, $C=1+h$ est une constante multiplicative qui est le multiplicateur moyen constaté permettant de passer du nombre de cas un jour donné au nombre de cas attendu le jour suivant. La simulation faite au soir du 6 mars 2020 donne $C\approx 1,4$, autrement dit, on constate entre le 25 février et le 6 mars qu’environ $h=0,4=40\%$ de cas supplémentaires sont observés chaque jour (par rapport au jour précédent).
Graphique des cas observés du 25 février au 6 mars et de l’estimation faite
Code Python
# Nombre de nouveaux cas pas jours - à partir du 25 février 2020
nouveaux = [2,4,20,19,43,30,61,21,73,138,190]
n = len(nouveaux)
# Période utilisée pour effectuer la régression linéaire
# dec = n-9 pour l'effectuer sur les 9 derniers jours
deb = n-9
# fin = 11 pour s'arrêter au 7 mars (date à partir de laquelle les malades
# avec de faibles symptomes ne sont plus testés)
# fin = n pour tenir compte tous les cas jusqu'aux derniers jours
fin = n
somcum = [12+sum(nouveaux[:(k+1)]) for k in range(n)]
# Nombre de jours pour lesquels on anticipe le nombre de personnes infectées
# en utilisant l'estimation exponentielle
dt = 1
# Le reste est automatique, ne pas y toucher
# (sauf si vous savez ce que vous faites ;-p)
import numpy as np
import matplotlib.pyplot as plt
import datetime as dtm
import matplotlib.dates as mdates
import matplotlib.patches as mpat
X = np.array(range(deb,fin))
Y = np.log(somcum)[deb:fin]
plt.plot(X,Y)
# Calcul des coefficients de la droite de régression
# ln(nb cas) = a*jour+b
moyX = sum(X)/(fin-deb)
moyY = sum(Y)/(fin-deb)
a = sum((X-moyX)*(Y-moyY))/sum((X-moyX)**2)
b = moyY-a*moyX
# Affichage de la droite de régression
plt.plot(X,a*X+b)
# Nouvelle figure et paramétrage
plt.figure()
ax = plt.gca()
formatter = mdates.DateFormatter("%d/%m")
ax.xaxis.set_major_formatter(formatter)
locator = mdates.DayLocator()
ax.xaxis.set_major_locator(locator)
# Histogrammes du nombre réel de cas testés positifs
dates = [dtm.datetime.fromordinal(737479+k) for k in range(n+1)]
dates = [str(k.day)+'/'+str(k.month)+'/'+str(k.year) for k in dates]
lbl = [dtm.datetime.strptime(d,"%d/%m/%Y").date() for d in dates]
for k,l in zip(somcum,lbl[:-1]):
m = mpat.Rectangle((l.toordinal()+0.5,0),1,k,color='red')
ax.add_patch(m)
plt.annotate(k, # this is the text
(l.toordinal()+1,k), # this is the point to label
textcoords="offset points", # how to position the text
xytext=(0,10), # distance from text to points (x,y)
ha='center', color='red')
# Affichage de la régression exponentielle
dates = [dtm.datetime.fromordinal(737480+k) for k in range(n+dt)]
dates = [str(k.day)+'/'+str(k.month)+'/'+str(k.year) for k in dates]
lbl = [dtm.datetime.strptime(d,"%d/%m/%Y").date().toordinal() for d in dates]
XX = range(n+dt)
YY = np.exp(a*XX+b)
plt.plot(lbl,YY,'-.k')
for k,l in zip(YY[-dt:],lbl[-dt:]):
plt.annotate(str(int(round(k)))+' ?', (l,int(k)),
textcoords="offset points", xytext=(0,-10),
ha='center', color='black')
# Légendes et autres labels sur la figure
plt.legend(["Régression exponentielle","Nombre de cas testés positifs"])
plt.title("Évolution du nombre de cas de coronavirus en France : testés positifs et estimés")
plt.xlabel("Date (2020)")
plt.ylabel('Nombre de cas')
# Affichage des données et des estimations
print("Nb de cas testés positifs du 25 février au",n-4,"mars 2020")
sc = [str(k) for k in somcum]
aff1 = [' : '.join(k) for k in zip(dates[:-dt],sc)]
sc2 = [str(k) for k in np.uint(YY[:-dt])]
aff2 = [' : '.join(k) for k in zip(dates[:-dt],sc2)]
print(*aff1,sep='\n')
print("Régression exponentielle du nombre de cas (même période)")
print(*aff2,sep='\n')
print("Estimation de nombre de cas à venir :")
print(dates[-dt],':',np.uint(np.round(YY[-dt])))
Régression sigmoïdale
On modélise cette fois le nombre de cas par une fonction du type
\[N_T=\dfrac{c}{1+e^{-(aT+b)}}\]Ceci équivaut à trouver trois réels $a,b,c$ tels que $aT+b\approx \ln\left(\frac{N_T}{c-N_T}\right)$. Le principe de l’algorithme présenté ci-dessous est d’obtenir, pour plusieurs valeurs de $c$ les coefficients $a,b$ en effectuant une régression linéaire classique sur $\ln\left(\frac{N_T}{c-N_T}\right)$, puis de choisir la valeur de $c$ minimisant la somme des carrés des différences entre données et résidus pour $T\mapsto\dfrac{c}{1+e^{-(aT+b)}}$.
Il est à noter que cette méthode ne peut être considérée comme vraiment satisfaisante dans la mesure où $a$ et $b$ sont obtenus par la méthode des moindres carrés appliquée à $\ln\left(\frac{N_T}{c-N_T}\right)$ tandis que $c$ minimise une autre fonctionnelle.
Graphique des cas observés du 25 février au 7 mars et de l’estimation faite pour le modèle sigmoïdal
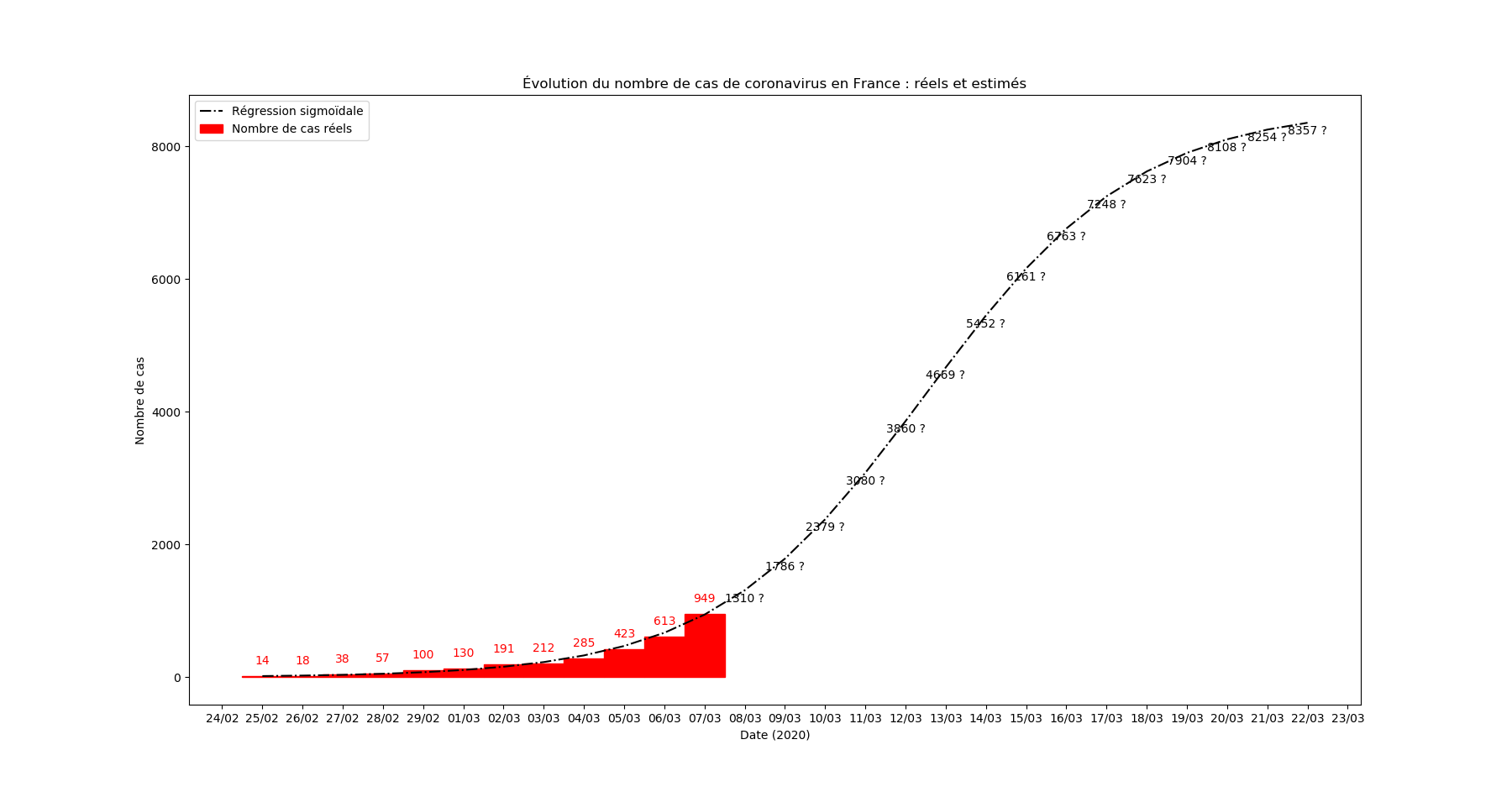
Si cette modélisation est valable, cela aurait pour conséquence que le nombre de cas plafonnerait aux environs de 9000 malades en France…
Attention ! Le SI est de taille en l’occurrence…
Variante Python
(mise-à–jour du code le 12 mars au matin)
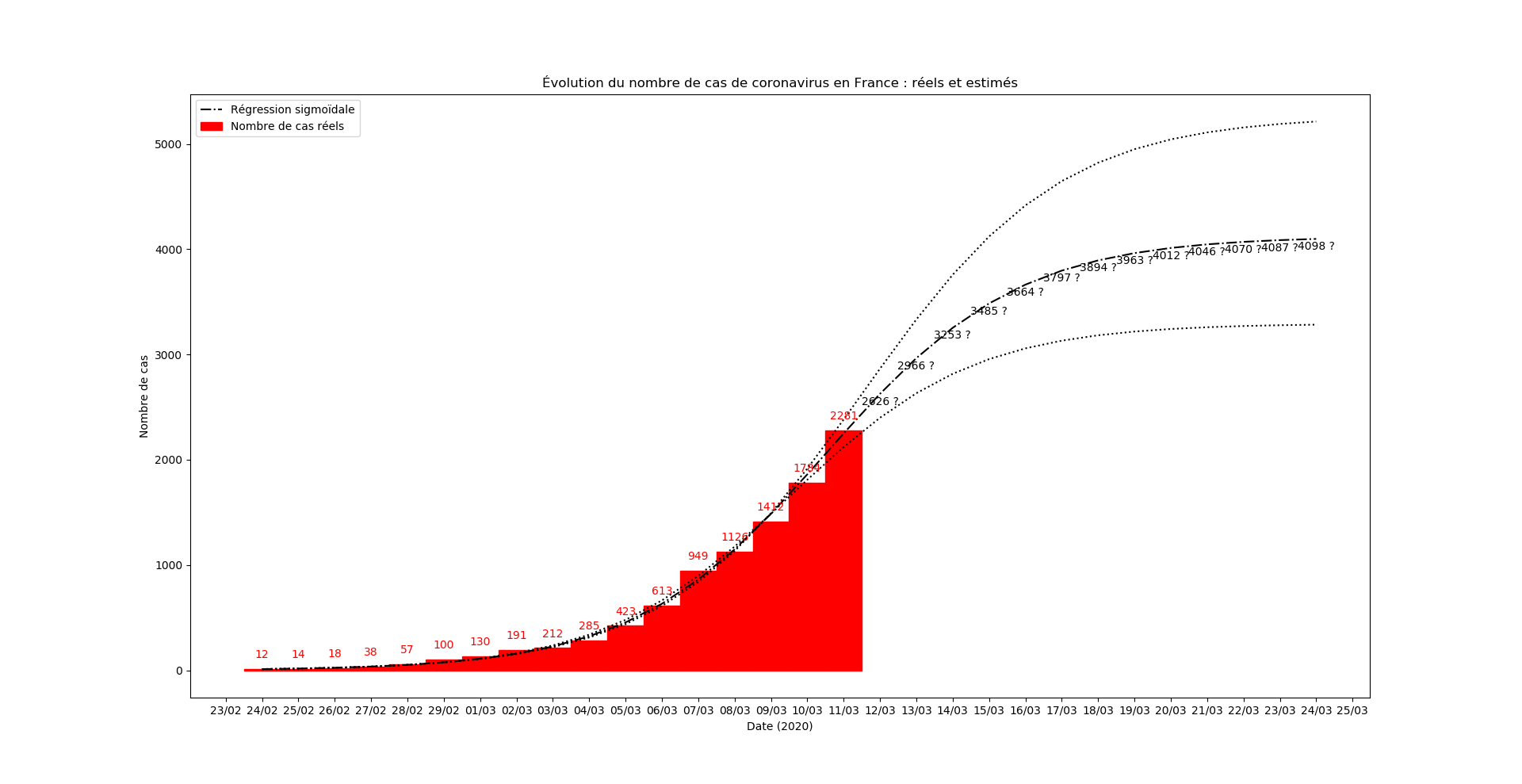
J’ai rajouté le tracé de deux sigmoïdes extrêmes qui approximent les données au pire deux fois moins précisément que la meilleure interpolation. Ce qui donne, pour les cas connus jusqu’au 11 mars, le graphique suivant :
# Nombre de nouveaux cas par jour - à partir du 25 février 2020
nouveaux = [0,2,4,20,19,43,30,61,21,73,138,190,336,177,286,372,497]
n = len(nouveaux)
# Période utilisée pour effectuer la régression linéaire
# deb = n-9 pour l'effectuer sur les 9 derniers jours
# deb = 0 pour l'effectuer à partir du 25 février
deb = 0
# fin = 13 pour s'arrêter au 7 mars (date à partir de laquelle les malades
# avec de faibles symptomes ne sont plus testés)
# fin = n pour tenir de compte de tous les cas jusqu'aux derniers jours
fin = n
somcum = [12+sum(nouveaux[:(k+1)]) for k in range(n)]
# Nombre de jours pour lesquels on anticipe le nombre de personnes infectées
# en utilisant l'estimation sigmoïdale
dt = 30-n
# Le reste est automatique, ne pas y toucher
# (sauf si vous savez ce que vous faites ;-p)
import numpy as np
import matplotlib.pyplot as plt
import datetime as dtm
import matplotlib.dates as mdates
import matplotlib.patches as mpat
def reglin(X,Y):
# Calcul des coefficients de la droite de régression
# Y = a*X+b
moyX = sum(X)/(fin-deb)
moyY = sum(Y)/(fin-deb)
a = sum((X-moyX)*(Y-moyY))/sum((X-moyX)**2)
b = moyY-a*moyX
return a,b
def f(a,b,c):
return lambda x:c/(1+np.exp(-(a*x+b)))
mn=np.infty
X = np.array(range(deb,fin))
tab = []
for c in np.exp(np.linspace(np.log(1+max(somcum)),np.log(6e7),500)):
Y0 = np.log(somcum/(c-somcum))[deb:fin]
a,b = reglin(X,Y0)
fX = f(a,b,c)(X)
S = sum((somcum[deb:fin]-fX)**2)
tab.append((a,b,c,S))
if S<=mn:
Y=Y0
am,bm,cm = a,b,c
mn = S
a,b,c = am,bm,cm
tab = [k for k in tab if k[3]<2*mn]
am,bm,cm = tab[0][:3]
aM,bM,cM = tab[-1][:3]
# affichage de X,Y
plt.plot(X,Y)
# Affichage de la droite de régression
plt.plot(X,a*X+b)
# Nouvelle figure et paramétrage
plt.figure()
ax = plt.gca()
formatter = mdates.DateFormatter("%d/%m")
ax.xaxis.set_major_formatter(formatter)
locator = mdates.DayLocator()
ax.xaxis.set_major_locator(locator)
# Histogrammes du nombre réel de cas testés positifs
dates = [dtm.datetime.fromordinal(737478+k) for k in range(n+1)]
dates = [str(k.day)+'/'+str(k.month)+'/'+str(k.year) for k in dates]
lbl = [dtm.datetime.strptime(d,"%d/%m/%Y").date() for d in dates]
for k,l in zip(somcum,lbl[:-1]):
m = mpat.Rectangle((l.toordinal()+0.5,0),1,k,color='red')
ax.add_patch(m)
plt.annotate(k, (l.toordinal()+1,k), textcoords="offset points",
xytext=(0,10), ha='center', color='red')
# Affichage de la régression sigmoïdale
dates = [dtm.datetime.fromordinal(737479+k) for k in range(n+dt)]
dates = [str(k.day)+'/'+str(k.month)+'/'+str(k.year) for k in dates]
lbl = [dtm.datetime.strptime(d,"%d/%m/%Y").date().toordinal() for d in dates]
XX = range(n+dt)
YY = f(a,b,c)(XX)
plt.plot(lbl,YY,'-.k')
for k,l in zip(YY[-dt-(n-fin):],lbl[-dt-(n-fin):]):
plt.annotate(str(int(round(k)))+' ?', (l,int(k)),
textcoords="offset points", xytext=(0,-10),
ha='center', color='black')
# Légendes et autres labels sur la figure
plt.legend(["Régression sigmoïdale","Nombre de cas testés positifs"])
plt.title("Évolution du nombre de cas de coronavirus en France : testés positifs et estimés")
plt.xlabel("Date (2020)")
plt.ylabel('Nombre de cas')
# Tracés des deux sigmoïdes extrémales approximant les données au pire deux fois
# moins bien que la meilleure interpolation
YY = f(am,bm,cm)(XX)
print(YY[-1])
plt.plot(lbl,YY,':k')
YY = f(aM,bM,cM)(XX)
print(YY[-1])
plt.plot(lbl,YY,':k')
# Affichage des données et des estimations
print("Nb de cas testés positifs du 24 février au",n-4,"mars 2020")
sc = [str(k) for k in somcum]
aff1 = [' : '.join(k) for k in zip(dates[:-dt],sc)]
sc2 = [str(k) for k in np.uint(YY[:-dt])]
aff2 = [' : '.join(k) for k in zip(dates[:-dt],sc2)]
print(*aff1,sep='\n')
print("Régression sigmoïdale du nombre de cas (même période)")
print(*aff2,sep='\n')
print("Estimation du nombre de cas à venir :")
print(dates[-dt],':',np.uint(np.round(YY[-dt])))
print("Paramètre sigmoïde :\nN=",format(int(c),'04d'),'/[1+exp(-(',format(a,'02f'),
'*T+',format(b,'02f'),'))]',sep='')
print("Taux d'évolution caractéristique du nombre de cas :",format(np.exp(a)-1,'.3f'))
day = n-1
taux = a*np.exp(-(a*day+b))/(1+np.exp(-(a*day+b)))
print("Taux d'évolution actuel du nombre de cas :",format(taux,'.3f'))
k = dtm.datetime.fromordinal(737479-int(round(b/a)))
print("Inflexion le :",str(k.day)+'/'+str(k.month)+'/'+str(k.year))
Graphique au 9 mars au soir
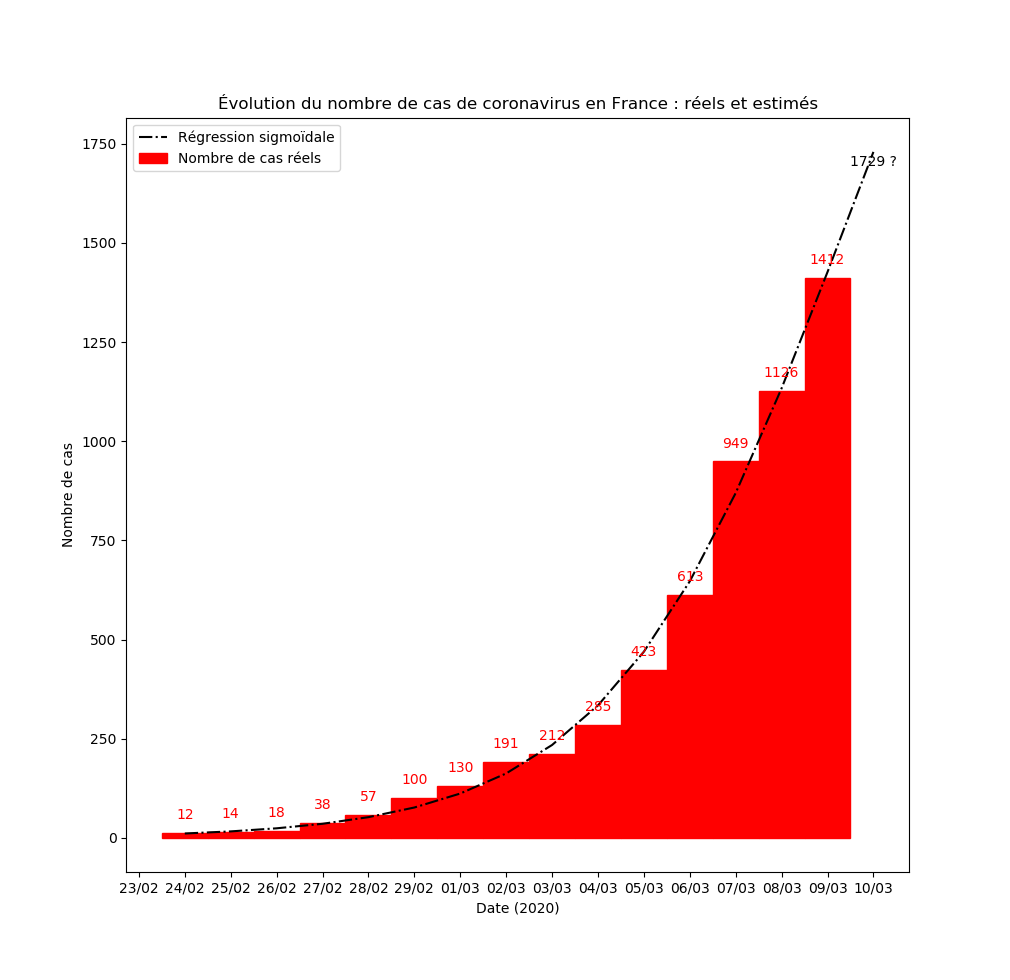
Une petite analyse des résultats actuels : si le modèle est valide, le nombre total de personnes atteintes en fin d’épidémie serait, en France, inférieur à 5000 et l’épidémie serait essentiellement terminée aux alentours du 25 mars 2020 (en France).
Pondération des données
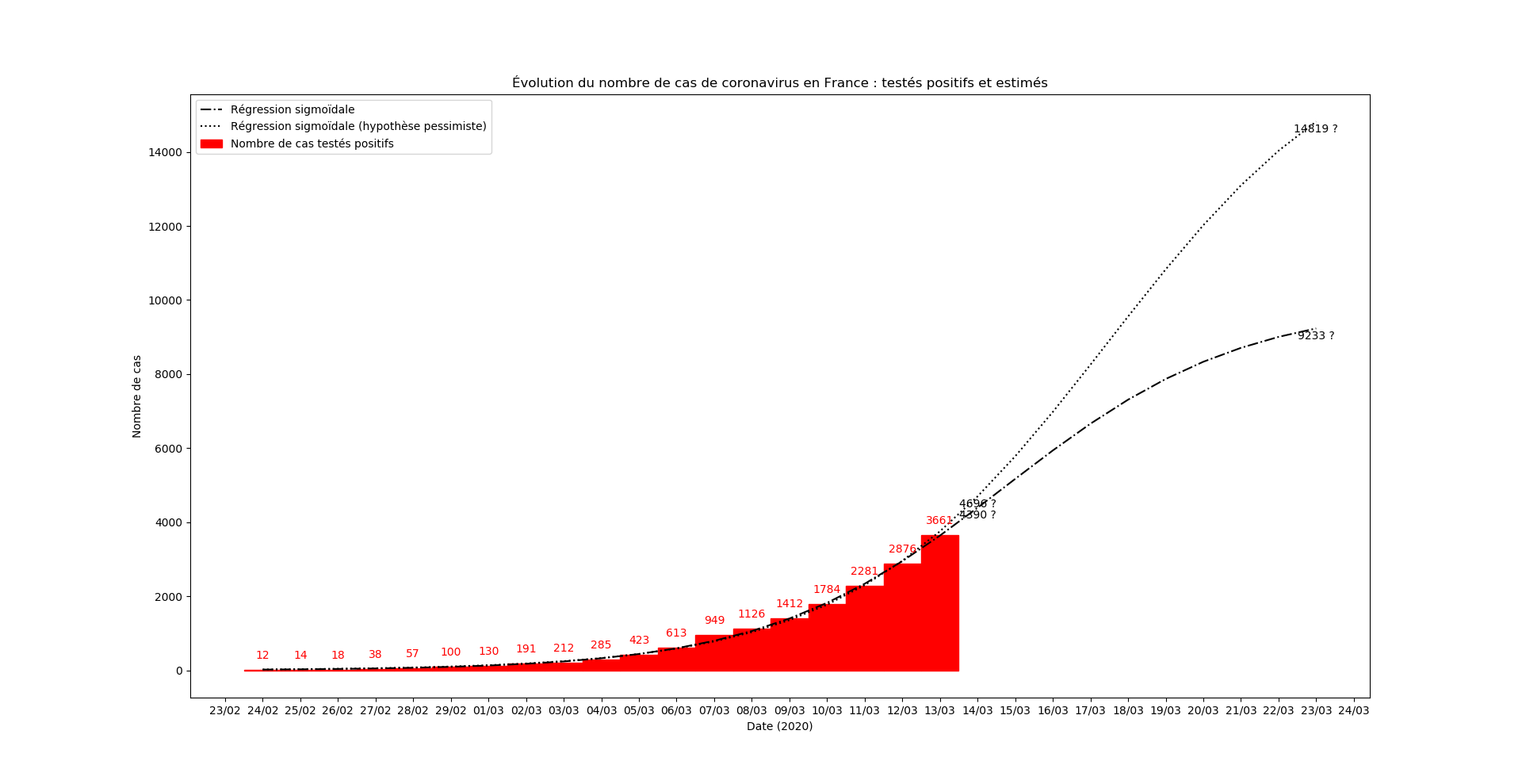
(mise-à-jour du 14 mars)
La précédente version de l’algorithme sous-estimait la croissance exponentielle de l’épidémie, notamment parce que l’optimisation des constantes $a$ et $b$ se fait par la méthode des moindres carrés appliquée au logarithme et non aux nombres de cas testés positifs. Pour atténuer cet effet, l’algorithme ci-dessous pondère les points sur lesquels la régression linéaire est effectuée par la population à laquelle ce point correspond. Ce nouvel algorithme se montre beaucoup plus robuste… et nettement moins optimiste.
import numpy as np
# Nombre de nouveaux cas par jour - à partir du 25 février 2020
nouveaux = [0,2,4,20,19,43,30,61,21,73,138,190,336,177,286,372,497,595,785]
n = len(nouveaux)
# Période utilisée pour effectuer la régression linéaire
# deb = n-9 pour l'effectuer sur les 9 derniers jours
# deb = 0 pour l'effectuer à partir du 25 février
deb = 0
# fin = 13 pour s'arrêter au 7 mars (date à partir de laquelle les malades
# avec de faibles symptomes ne sont plus testés)
# fin = n pour tenir de compte de tous les cas jusqu'aux derniers jours
fin = n
somcum = np.array([12+sum(nouveaux[:(k+1)]) for k in range(n)])
# Nombre de jours pour lesquels on anticipe le nombre de personnes infectées
# en utilisant l'estimation sigmoïdale
dt = 10
# Le reste est automatique, ne pas y toucher
# (sauf si vous savez ce que vous faites ;-p)
import matplotlib.pyplot as plt
import datetime as dtm
import matplotlib.dates as mdates
import matplotlib.patches as mpat
def reglin(X,Y,poids=None):
# Calcul des coefficients de la droite de régression
# Y = a*X+b
if poids is None:
poids=np.ones((len(X),))
poids = np.array(poids)
N = np.sum(poids)
moyX = sum(poids*X)/N
moyY = sum(poids*Y)/N
a = sum(poids*(X-moyX)*(Y-moyY)/N)/sum(poids*(X-moyX)**2/N)
b = moyY-a*moyX
return a,b
def f(a,b,c):
return lambda x:c/(1+np.exp(-(a*x+b)))
mn=np.infty
X = np.array(range(deb,fin))
tab = []
for c in np.exp(np.linspace(np.log(1+max(somcum[deb:fin])),np.log(7e7),5000)):
Y0 = np.log(somcum/(c-somcum))[deb:fin]
a,b = reglin(X,Y0,somcum)
fX = f(a,b,c)(X)
S = sum((somcum[deb:fin]-fX)**2)
tab.append((a,b,c,S))
if S<=mn:
Y=Y0
am,bm,cm = a,b,c
mn = S
a,b,c = am,bm,cm
tab = [k for k in tab if k[3]<1.5*mn]
am,bm,cm = tab[0][:3]
aM,bM,cM = tab[-1][:3]
# affichage de X,Y
plt.plot(X,Y)
# Affichage de la droite de régression
plt.plot(X,a*X+b)
# Nouvelle figure et paramétrage
plt.figure()
ax = plt.gca()
formatter = mdates.DateFormatter("%d/%m")
ax.xaxis.set_major_formatter(formatter)
locator = mdates.DayLocator()
ax.xaxis.set_major_locator(locator)
# Histogrammes du nombre réel de cas testés positifs
dates = [dtm.datetime.fromordinal(737478+k) for k in range(n+1)]
dates = [str(k.day)+'/'+str(k.month)+'/'+str(k.year) for k in dates]
lbl = [dtm.datetime.strptime(d,"%d/%m/%Y").date() for d in dates]
for k,l in zip(somcum,lbl[:-1]):
m = mpat.Rectangle((l.toordinal()+0.5,0),1,k,color='red')
ax.add_patch(m)
plt.annotate(k, (l.toordinal()+1,k), textcoords="offset points",
xytext=(0,10), ha='center', color='red')
# Affichage de la régression sigmoïdale
dates = [dtm.datetime.fromordinal(737479+k) for k in range(n+dt)]
dates = [str(k.day)+'/'+str(k.month)+'/'+str(k.year) for k in dates]
lbl = [dtm.datetime.strptime(d,"%d/%m/%Y").date().toordinal() for d in dates]
XX = range(n+dt)
YY = f(a,b,c)(XX)
plt.plot(lbl,YY,'-.k')
for k,l in zip(YY[-dt-(n-fin)::dt+n-fin-1],lbl[-dt-(n-fin)::dt+n-fin-1]):
plt.annotate(str(int(round(k)))+' ?', (l,int(k)),
textcoords="offset points", xytext=(0,-10),
ha='center', color='black')
# Légendes et autres labels sur la figure
plt.title("Évolution du nombre de cas de coronavirus en France : testés positifs et estimés")
plt.xlabel("Date (2020)")
plt.ylabel('Nombre de cas')
YY2 = f(aM,bM,cM)(XX)
print(YY2[-1])
plt.plot(lbl,YY2,':k')
plt.annotate(str(int(round(YY2[-1])))+' ?', (lbl[-1],int(YY2[-1])),
textcoords="offset points", xytext=(0,-10),
ha='center', color='black')
plt.annotate(str(int(round(YY2[-dt-n+fin])))+' ?', (lbl[-dt-n+fin],int(YY2[-dt-n+fin])),
textcoords="offset points", xytext=(0,-10),
ha='center', color='black')
plt.legend(["Régression sigmoïdale","Régression sigmoïdale (hypothèse pessimiste)","Nombre de cas testés positifs"])
# Affichage des données et des estimations
YY = f(a,b,c)(XX)
print("Nb de cas testés positifs du 24 février au",n-4,"mars 2020")
sc = [str(k) for k in somcum]
aff1 = [' : '.join(k) for k in zip(dates[:-dt],sc)]
sc2 = [str(k) for k in np.uint(YY[:-dt])]
aff2 = [' : '.join(k) for k in zip(dates[:-dt],sc2)]
print(*aff1,sep='\n')
print("Régression sigmoïdale du nombre de cas (même période)")
print(*aff2,sep='\n')
print()
print("Estimation du nombre de cas à venir :")
print(dates[-dt],':',np.uint(np.round(YY[-dt])))
print("Paramètre sigmoïde :\nN=",format(int(c),'04d'),'/[1+exp(-(',format(a,'02f'),
'*T+',format(b,'02f'),'))]',sep='')
print("Intervalles :",(am,aM),(bm,bM),(cm,cM))
print("Taux d'évolution caractéristique du nombre de cas :",format(np.exp(a)-1,'.3f'))
day = n-1
taux = a*np.exp(-(a*day+b))/(1+np.exp(-(a*day+b)))
print("Taux d'évolution actuel du nombre de cas :",format(taux,'.3f'))
k = dtm.datetime.fromordinal(737479-int(round(b/a)))
print("Inflexion le :",str(k.day)+'/'+str(k.month)+'/'+str(k.year))
k = dtm.datetime.fromordinal(737479-int(round(b/a))*2-5)
print("Epidémie essentiellement terminée :\n",str(k.day)+'/'+str(k.month)+'/'+str(k.year))
print()
print("Estimation pessimiste du nombre de cas à venir :")
nouvdat = np.uint(np.round(YY2[-dt]))
print(dates[-dt],':',nouvdat)
print("Paramètre sigmoïde pessimiste :\nN=",format(int(cM),'04d'),'/[1+exp(-(',format(aM,'02f'),
'*T+',format(bM,'02f'),'))]',sep='')
print("Taux d'évolution pessimiste caractéristique du nombre de cas :",format(np.exp(aM)-1,'.3f'))
day = n-1
taux = aM*np.exp(-(aM*day+bM))/(1+np.exp(-(aM*day+bM)))
print("Taux d'évolution pessimiste actuel du nombre de cas :",format(taux,'.3f'))
k = dtm.datetime.fromordinal(737479-int(round(bM/aM)))
print("Inflexion pessimiste le :",str(k.day)+'/'+str(k.month)+'/'+str(k.year))
k = dtm.datetime.fromordinal(737479-int(round(bM/aM))*2-5)
print("Epidémie essentiellement terminée (hyp.pessimiste) :\n",str(k.day)+'/'+str(k.month)+'/'+str(k.year))